Epigenetik
Epigenetik ist definiert als vererbbare Veränderungen der Genexpression, die im Gegensatz zu Mutationen nicht auf Veränderungen der DNA-Sequenz zurückzuführen sind. Die vorherrschenden epigenetischen Mechanismen sind DNA-Methylierung, Modifikationen am Chromatin, Verlust der Prägung und nicht-kodierende RNA. Die epigenetische Regulierung der Genexpression scheint langfristige Effekte und weitreichende Auswirkungen auf die Gesundheit zu haben.1 Ein besseres Verständnis des Epigenoms ist sowohl für die Grundlagenforschung als auch für die Entwicklung von neuen Therapieansätzen von großer Relevanz.
Ein großer Teil der Epigenetik wird durch posttranslationale Modifikationen an Proteinen gesteuert. Methylierungen, Acetylierungen, Ubiquitinierungen oder Phosphorylierungen führen zu Veränderungen, die zum Beispiel die Transkription beeinflussen. Epigenetische Modifikationen finden häufig im Laufe des Lebens eines Organismus statt; diese Veränderungen können jedoch auf die nächste Generation übertragen werden, wenn sie in Keimzellen auftreten. Paramutation, Bookmarking, Imprinting, Gen-Silencing, X-Chromosom-Inaktivierung, Positionseffekt, veränderbare Störung oder phänotypischer Schweregrad, Reprogrammierung, mütterliche Eigenschaften, karzinogene Prozesse, teratogene Effekte, Regulation von Histonmodifikationen, Heterochromatin-Zustände und Klonen sind als epigenetische Prozesse bekannt.2
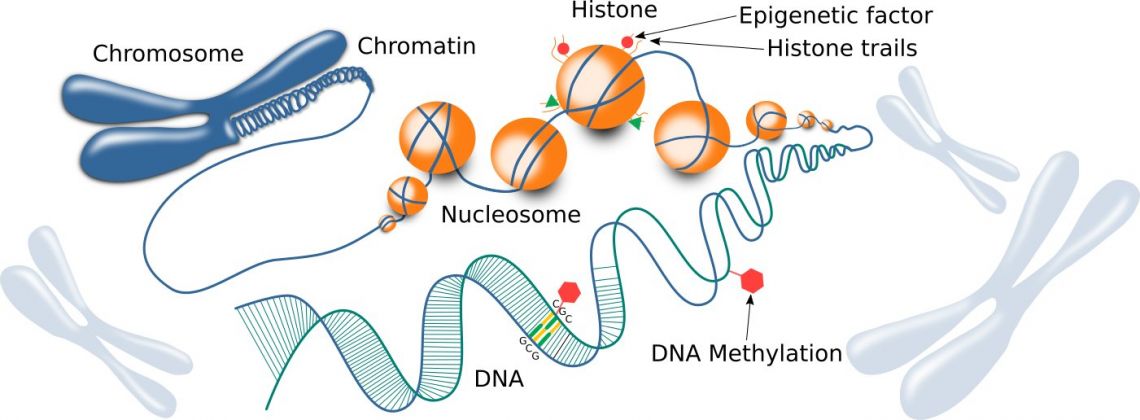
Abbildung 1: Chromatin-Struktur, einschließlich Histone und DNA.
DNA-Modifikationen
Die kovalente Bindung einer Methylgruppe an die C5-Position von Cytosin ist die wichtigste epigenetische Modifikation der DNA. Das Vorhandensein eines methylierten Cytosins kann die Transkription unterdrücken, indem es die Bindung von Transkriptionsfaktoren hemmt, oder die Bindung anderer Transkriptionsrepressoren fördern, einschließlich Histon-modifizierender Proteine wie Histon-Deacetylasen.3
DNA-Demethylierung spielt eine wichtige Rolle bei der Entwicklung und Tumorentstehung in Säugetieren. DNA-Demethylierung, die in primordialen Keimzellen und in frühen Embryonen auftritt, ist essentiell für Zellen, um in einen pluripotenten Zustand zurückzukehren.4
Histon-Modifikationen
Gleich der DNA-Methylierung beeinflussen posttranslationale Histon-Modifikationen nicht die DNA-Nukleotidsequenz, können aber deren Verfügbarkeit für die Transkriptionsmaschinerie modifizieren. Es sind mehrere Arten von Histonmodifikationen bekannt, von denen Acetylierung, Methylierung, Phosphorylierung und Ubiquitinierung die am besten untersuchten und wichtigsten in Bezug auf die Regulation der Chromatinstruktur und (Transkriptions-)Aktivität sind. 5 Während die DNA-Methylierung als eine sehr stabile epigenetische Modifikation gilt, sind die Histon-Modifikationen vergleichsweise labiler, wobei ihre Spiegel durch ein Gleichgewicht zwischen histonmodifizierenden Enzymen aufrechterhalten werden, die spezifische Modifikationen hinzufügen oder entfernen. Abweichende Histon-Modifikationsniveaus resultieren aus einem Ungleichgewicht dieser modifizierenden Enzyme in krankem Gewebe, so dass die Korrektur des erhöhten oder verminderten Niveaus eines bestimmten Enzyms das natürliche Gleichgewicht in den betroffenen Zellen wiederherstellt.6
Chromatin-Umbau
Das Vorhandensein von Histonen wirkt wie eine Barriere für den Zugang zu Proteinen; daher muss für essentielle Prozesse wie Transkription und Replikation ein Chromatin-Remodeling stattfinden. In Verbindung mit Histon-Modifikationen spielt die DNA-Methylierung eine entscheidende Rolle beim Gen-Silencing durch Chromatin-Remodeling. Chromatin-Remodeling ist auch mit der DNA-Schadensreaktion, der Aufrechterhaltung von Stammzelleigenschaften und Zelldifferenzierungsprogrammen verknüpft. Es hat sich zunehmend gezeigt, dass Chromatinmodifikationen zu lang anhaltenden Veränderungen der Chromatinstruktur und der Transkription führen. Neuere Studien haben gezeigt, dass Umwelteinflüsse in utero das Potenzial haben, normale Entwicklungssignalnetzwerke, physiologische Reaktionen und Krankheitsanfälligkeit im späteren Leben während eines Prozesses zu verändern, der als Entwicklungsreprogrammierung bekannt ist.7
Transkriptionsfaktoren
Chemische Modifikationen von Histonen und DNA, wie Histon-Methylierung, Histon-Acetylierung und DNA-Methylierung, spielen eine entscheidende Rolle bei der epigenetischen Genregulation. Viele der Enzyme, die solche chemischen Modifikationen hinzufügen oder entfernen, sind dafür bekannt, oder es wird vermutet, dass sie empfindlich auf Veränderungen im intrazellulären Stoffwechsel reagieren. Sie nutzen (oft) Kosubstrate, die durch den zellulären Stoffwechsel erzeugt werden, und stellen damit eine mögliche Verbindung zwischen Ernährung, Stoffwechsel und Genregulation her.8
Populäre Epigenetics Antibodies
- (1)
- (9)
- (1)
- (8)
- (14)
- (1)
- (7)
- (20)
- (1)
- (4)
- (21)
- (1)
- (4)
- (1)
- (4)
- (5)
- (1)
- (3)
- (5)
- (1)
- (1)
- (1)
- (1)
- (3)
Brauchen Sie Hilfe? Rufen Sie unseren PhD-Kundensupport an!
- Wir helfen Ihnen bei der Suche nach dem richtigen Produkt für Ihre Forschung.
- Wir bieten zuverlässige Antikörper, Kits, Proteine, Lysate für Ihre Forschung.
- Kontaktieren Sie uns per E-Mail oder Telefon: (877) 302 8632 (US) oder +49 241 95 163 153 (International)
Verwandte Informationen und Produkte
Einführung in die Chromatin-Immunopräzipitation (ChIP)
CUT&RUN - Verbesserte Methode zur Untersuchung von Protein-DNA-Interaktionen
Referenzen
- : "Epigenetics: principles and practice." in: Digestive diseases (Basel, Switzerland), Vol. 29, Issue 2, pp. 130-5, (2011) (PubMed).
- : "Role of Epigenetics in Biology and Human Diseases." in: Iranian biomedical journal, Vol. 20, Issue 5, pp. 246-58, (2017) (PubMed).
- : "Epigenetic modifications: basic mechanisms and role in cardiovascular disease (2013 Grover Conference series)." in: Pulmonary circulation, Vol. 4, Issue 2, pp. 169-74, (2014) (PubMed).
- : "DNA methylation: superior or subordinate in the epigenetic hierarchy?" in: Genes & cancer, Vol. 2, Issue 6, pp. 607-17, (2011) (PubMed).
- : "Histone modifications and their role in epigenetics of atopy and allergic diseases." in: Allergy, asthma, and clinical immunology : official journal of the Canadian Society of Allergy and Clinical Immunology, Vol. 14, pp. 39, (2018) (PubMed).
- : "Epigenetic modifications as therapeutic targets." in: Nature biotechnology, Vol. 28, Issue 10, pp. 1069-78, (2015) (PubMed).
- : "Long-term effects of chromatin remodeling and DNA damage in stem cells induced by environmental and dietary agents." in: Journal of environmental pathology, toxicology and oncology : official organ of the International Society for Environmental Toxicology and Cancer, Vol. 32, Issue 4, pp. 307-27, (2014) (PubMed).
- : "Influence of metabolism on epigenetics and disease." in: Cell, Vol. 153, Issue 1, pp. 56-69, (2013) (PubMed).




