SARS-CoV-2 Neutralisierende Antikörper
Neutralisierende Antikörper sind für Wissenschaftler von besonderem Interesse. Sie stoppen die Infektion effizient, indem sie die Interaktion zwischen dem SARS-CoV-2-Virus und den Wirtszellen blockieren. Die meisten neutralisierenden Antikörper reagieren auf die Rezeptorbindungsdomäne (RBD) des Spike-Proteins, das direkt an den Zelloberflächenrezeptor ACE2 bindet. antibodies-online.com bietet zwei nAbs auf Basis des Klons CR3022 sowie zwei nAbs auf Basis des Klons MM43 an.
Alle neutralisierenden SARS-CoV-2-Antikörper anzeigen
Eine der sich abzeichnenden Fragen im Zusammenhang mit neu auftauchenden Varianten von SARS-CoV-2 ist, ob bestehende Antikörper-basierte Assays und Pharmazeutika weiterhin verwendet werden können. In Zusammenarbeit mit Nanotemper haben wir die Affinität unseres S-Protein-RBD-Antikörpers CR3022 (ABIN6952546) zum kanonischen trimeren SARS-CoV-2 S-Protein und zu den Varianten B.1.1.7 / alpha, B.1.351 / beta, B.1.617.2 / delta, B.1.617.1 / kappa und P.1 / gamma mittels mikroskaliger Thermophorese (MST) gemessen. In einer zweiten Iteration erkannten wir die dominante B.1.1.529 / omicron-Variante an und erweiterten die Antikörperauswahl mit neutralisierenden Antikörpern MM43 (ABIN7036075) und MM117 (ABIN7042145) erweitert. Entdecken Sie die Ergebnisse weiter unten, entweder zusammengefasst in der Tabelle oder in ausführlicher Form mit angehängten MST-Bindungskurven. Unser Webinar bietet weitere Hintergrundinformationen zu den Experimenten und den Auswirkungen auf die Tests und die Impfstoffproduktion. Wir bemühen uns, die Vergleichsmessungen im Hinblick auf neue Antikörper und aufkommende Varianten auf dem neuesten Stand zu halten.
MST-Analyse von SARS-CoV-2 nAbs
Webinar: "Effect of SARS-CoV-2 Variants for Assays and Vaccines"
SARS-CoV-2 neutralisierende Antikörper und zugehörige Bindungsaffinitäten
| Canonical | Delta B.1.617.2 | Omicron B.1.1.529 | |
|---|---|---|---|
| CR3022 ABIN6952546 |
++ | + | + |
| MM43 |
+++ | ++ | - |
| MM117 |
+ | + | ++ |
Microscale Thermophorese-Analyse von SARS-CoV-2 nAbs
CR3022
Der Antikörperklon CR3022 (ABIN6952546), der die Grundlage für beide Antikörper bildet, wurde ursprünglich von einem rekonvaleszenten SARS-Patienten aus Singapur isoliert 1. Der Klon erwies sich in Neutralisierungstests für verschiedene SARS-CoV-Stämme in Synergie mit anderen RBD-gerichteten Antikörpern als wirksam. Sein Epitop überschneidet sich nicht mit der Bindungsstelle des Angiotensin-konvertierenden Enzyms 2 (ACE2) und ist somit für andere neutralisierende Antikörper zugänglich. Seit dem Ausbruch von COVID-19 wurde nachgewiesen, dass CR3022 das SARS-CoV-2 S-Protein RBD auf ähnliche Weise bindet2. Kristallisationsuntersuchungen von CR3022, das an sein SARS-CoV-2-Target gebunden ist, haben wichtige Erkenntnisse über mögliche Angriffspunkte für Therapeutika gegen dieses Virus geliefert 3. Darüber hinaus wurde CR3022 als Positivkontrolle in serologischen Tests zum Nachweis von Antikörpern in Humanserum verwendet, die das SARS-CoV-2 S-Protein binden4.
Eine höhere Dissoziationskonstante deutet auf eine schwächere Bindung hin, d.h. die Bindung von CR3022 an das B.1.351 S-Protein ist deutlich schwächer als bei den anderen drei Varianten. Dennoch deutet eine KD im nanomolaren Bereich auf eine hohe Affinität des Antikörpers zu allen vier getesteten S-Proteinen hin. Somit bleibt der neutralisierende Antikörper ein wichtiges Werkzeug für SARS-CoV-2-Immunoassays.
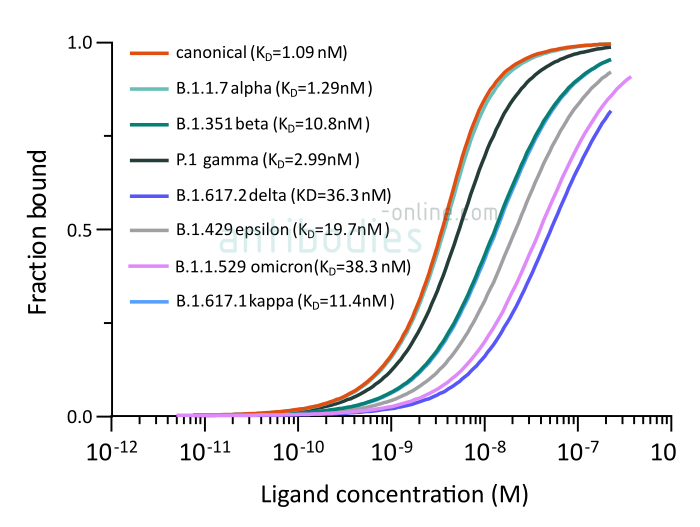
Fig. 1: Thermophorese-Messungen im Mikrobereich zur Bindung des SARS-CoV-2 Spike S1-Antikörpers (RBD) CR3022 (ABIN6952546) an SARS-CoV-2 Spike (Trimer) von wt SARS-CoV-2 (ABIN6952670) oder Varianten B.1 .1.7 alpha (ABIN6963742), B.1.351 beta (ABIN6963740), P.1 gamma (ABIN6964443), B.1.6172 delta (ABIN6999328), B.1.429 epsilon (ABIN6972926), B.1.617.1 kappa (ABIN6976302) and B.1.1.529 omicron (ABIN7041433). Die ermittelten Dissoziationskonstanten KD sind angegeben.
MM117
MM117 (ABIN7042145) ist ein in Mäusen gezüchteter monoklonaler Antikörper, der das SARS-CoV-2 B.1.1.529 Omicron-S-Protein stärker bindet als andere Varianten.
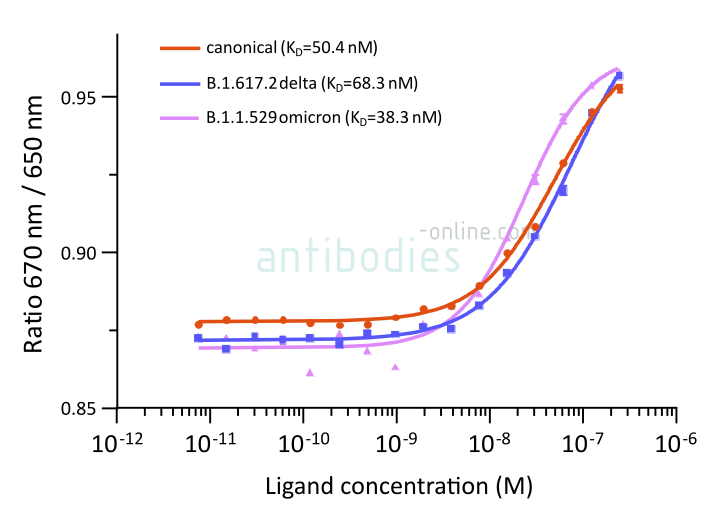
Fig. 2: Mikroskalige Thermophorese-Messungen der Bindung des SARS-CoV-2 Spike S1-Antikörpers (RBD) MM117 (ABIN7042145) an SARS-CoV-2 Spike (Trimer)-Proteine verschiedener Linien, einschließlich B.1.617.2 (delta) und B.1.1.529 (omicron). Die ermittelten Dissoziationskonstanten KD sind angegeben.
MM43
Der monoclonale Antikörper MM43 (ABIN7036075) hingegen binden das kanonische S-Protein fester als CR3022 und MM117. Die Bindung an das SARS-CoV-2 B.1.617.2 delta S-Protein ist ebenfalls sehr gut. Die Bindung dieses Antikörpers an das S-Protein der Omicron-Variante ist jedoch aufgrund von Mutationen des Proteins vollständig aufgehoben.
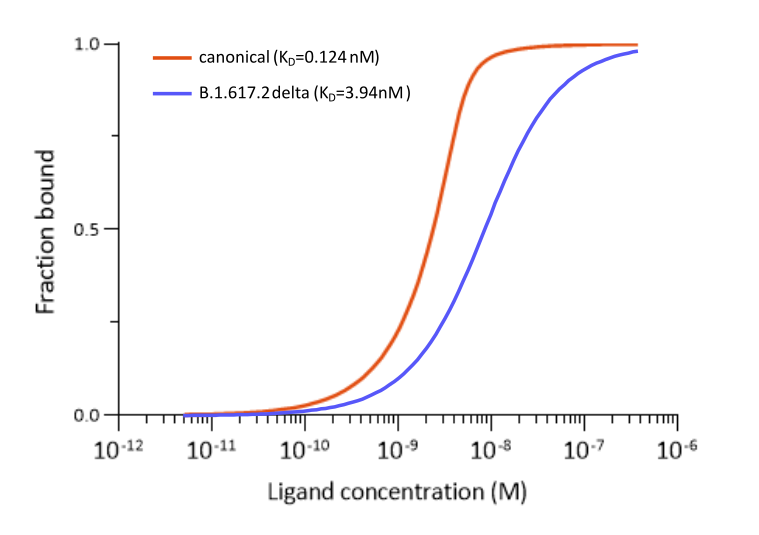
Abb. 3: Mikroskalige Thermophorese-Messungen der Bindung des SARS-CoV-2 Spike S1-Antikörpers (RBD) MM43 (ABIN7036075) an SARS-CoV-2 Spike (Trimer) verschiedener Linien. Bindet SARS-CoV-2 S-Proteine verschiedener Linien, einschließlich B.1.617.2 (delta). Bindet das S-Protein von SARS-CoV-2 B.1.1.529 (omicron) nicht. Die ermittelten Dissoziationskonstanten KD sind angegeben.
Verfügbare SARS-CoV-2 neutralisierende Antikörper
- (8)
- (15)
- (1)
- (5)
- (3)
- (1)
Gedankenexperiment vor der MST-Messung
Das SARS-CoV-2 S-Protein ist essentiell für den Kontakt der Virionen mit der Wirtszelle über den ACE2-Rezeptor. Die Kontaktpunkte von ACE2 befinden sich zwischen den Aminosäuren K417 und Y505 der S-Protein-Rezeptorbindungsdomäne (RBD). Neutralisierende Antikörper (nAbs), die auf die RBD abzielen, unterbrechen diese Interaktion und können so die Interaktion zwischen SARS und CoV-2 verhindern. Aufgrund der bekannten Kontaktpunkte des neutralisierenden Antikörpers CR3022 mit dem S-Protein hatten wir vorhergesagt, dass der Antikörper das Protein auch bei bedenklichen Varianten binden würde (Abb. 4)6,7,8,9.
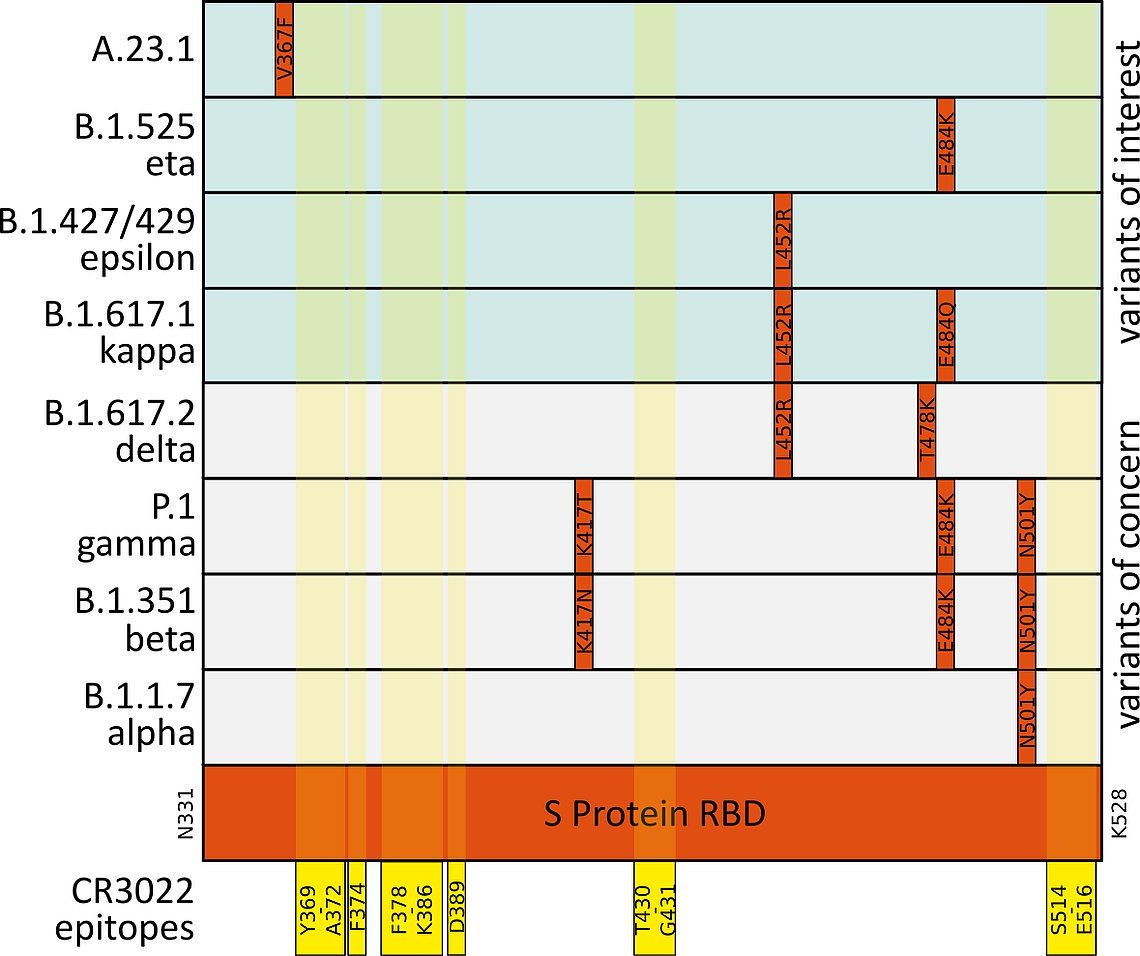
Fig. 4: Mutations ( rot gekennzeichnet) in der SARS-CoV-2 S-Protein Rezeptorbindungsdomäne (RBD, mint) in bedenklichen (hellgrau) und unbedenklichen (hellmint) Varianten. Die Epitope des neutralisierenden Antikörpers CR3022 ABIN6952546 sind gelb hervorgehoben.6,7,8,9
Diese Vorhersage wurde durch die MST-Messungen bestätigt. Die Variante B.1.1.529 / omicron weist im Vergleich dazu die meisten Mutationen in der RBD-Stelle auf. Dennoch konnte die Bindung von CR3022 experimentell bestätigt werden. Vertiefende Hintergrundinformationen finden Sie in unserem Webinar "Effect of Different SARS-CoV-2 Variants on COVID-19 Assays and Vaccines".
Webinar: "Effect of Different SARS-CoV-2 Variants on COVID-19 Assays and Vaccines"
Referenzen
- (1) ter Meulen, J. et al. Human monoclonal antibody combination against SARS coronavirus: synergy and coverage of escape mutants. PLoS Med. 3, e237–e237 (2006).
- (2) Tian, X. et al. Potent binding of 2019 novel coronavirus spike protein by a SARS coronavirus-specific human monoclonal antibody. Emerg. Microbes Infect. 9, 382–385 (2020).
- (3) Yuan, M. et al. A highly conserved cryptic epitope in the receptor-binding domains of SARS-CoV-2 and SARS-CoV. Science 80. eabb7269 . doi:10.1126/science.abb7269 (2020).
- (4) Stadlbauer, D. et al. SARS-CoV-2 Seroconversion in Humans: A Detailed Protocol for a Serological Assay, Antigen Production, and Test Setup. Curr. Protoc. Microbiol. 57, e100 (2020).
- (5) Thao, T. et al. Rapid reconstruction of SARS-CoV-2 using a synthetic genomics platform Nature. (2020).
- (6) Barnes CO et al. SARS-CoV-2 neutralizing antibody structures inform therapeutic strategies. Nature (2020). doi:10.1038/s41586-020-2852-1
- (7) Cheng MH et al. Impact of South African 501.V2 Variant on SARS-CoV-2 Spike Infectivity and Neutralization: A Structure-based Computational Assessment. bioRxiv 2021.01.10.426143 (2021). doi:10.1101/2021.01.10.426143
- (8) Garcia-Beltran WF et al. Circulating SARS-CoV-2 variants escape neutralization by vaccine-induced humoral immunity. medRxiv 2021.02.14.21251704 (2021). doi:10.1101/2021.02.14.21251704
- (9) Yuan M et al. A highly conserved cryptic epitope in the receptor binding domains of SARS-CoV-2 and SARS-CoV. Science 368, 630–633 (2020).
- (10) Taylor, P.C., Adams, A.C., Hufford, M.M. et al. Neutralizing monoclonal antibodies for treatment of COVID-19. Nat Rev Immunol (2021). https://doi.org/10.1038/s41577-021-00542-x
- (11) Emily Engelhart, Randolph Lopez, Ryan Emerson, Charles Lin, Colleen Shikany, Daniel Guion, Mary Kelley, David Younger. Massively Multiplexed Affinity Characterization of Therapeutic Antibodies Against SARS-CoV-2 Variants. Preprint. bioRxiv 2021.04.27.440939; doi: https://doi.org/10.1101/2021.04.27.440939





