SARS-CoV-2 Interferon Antagonismus
Jüngste Studien über SARS-CoV-2, die die Hemmung der Immunantwort untersuchen, enthüllen mehrere SARS-CoV-2-Proteine, welche die Wirtsreaktion zugunsten der Virusvermehrung manipulieren. Im Vergleich zu anderen virusbedingten Atemwegserkrankungen wird das IFN-I- und IFN-III-Signal recht wirksam unterdrückt. Dies führt letztlich zu Reaktionen mit niedrigen Interferon-stimulierten Genen (ISG) mit Auswirkungen auf die Virusübertragung und Pathogenese.1
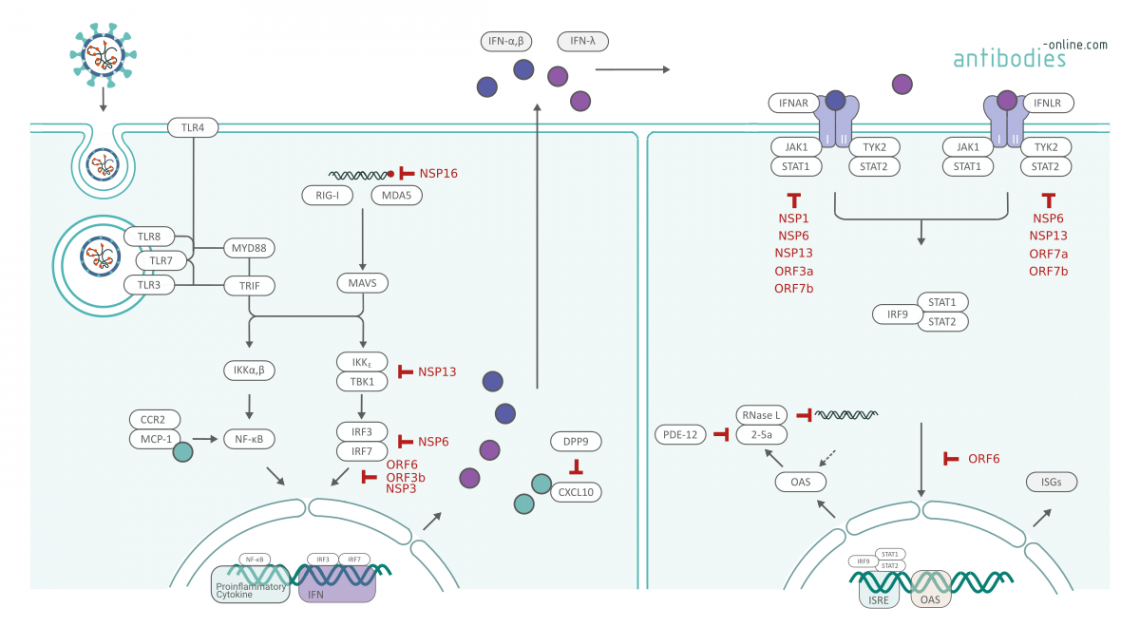
Interferon Antagonismus von SARS-Cov-2 Proteinen in Anlehnung an Xia et al.2
Interferon Antagonismus Antibodies
- (1)
- (8)
- (2)
- (4)
- (2)
- (3)
- (1)
- (1)
- (5)
- (6)
- (6)
- (1)
- (2)
- (3)
- (1)
SARS-CoV-2 antagonisiert IFN-I-Produktion und Signaling
Ein unbiased Screening der SARS-CoV-2-Proteine identifizierte den Hauptantagonisten der Typ-I-Interferon (IFN-I)-Reaktion.2 Drei SARS-CoV-2-Proteine antagonisieren die IFN-I-Produktion über unterschiedliche Mechanismen: Nichtstrukturprotein 6 (nsp6) bindet die TANK-bindende Kinase 1 (TBK1), um die Phosphorylierung des Interferon-Regulationsfaktors 3 (IRF3) zu unterdrücken, nsp13 bindet und blockiert die TBK1-Phosphorylierung, und das open reading frame 6 (ORF6) bindet das Importin Karyopherin α 2 (KPNA2), um die IRF3-Kerntranslokation zu hemmen.
Zwei Sets von viralen Proteinen antagonisieren das IFN-I-Signaling und IFN-III (IL28a/IL28b/IL29) Signaling durch Blockade des Signaltransduktors und des Aktivators der Transkription 1 (STAT1)/STAT2-Phosphorylierung beziehungsweise der Kerntranslokation. NSP1, ORF3a und ORF7b antagonisieren den STAT1-Signalweg, ORF7a und ORF7b antagonisieren den STAT2-Signalweg2. nsp6 und nsp13 sind Schlüsselakteure des Interferon-Antagonismus. Sie unterdrücken beide Signalwege zusammen mit der nuklearen Translokation von IRF3. Ähnlich wie bei SARS-CoV und MERS-CoV sind die Hauptsupressoren für SARS-CoV-2 nsp1 und nsp6; allerdings hemmt SARS-CoV-2 die Interferon-IFN-I-Signalübertragung effizienter.
Interferon Antagonism related Antibodies
- (3)
- (6)
- (2)
- (7)
- (8)
- (16)
- (3)
- (1)
- (2)
- (2)
- (2)
- (1)
- (3)
- (2)
- (4)
- (1)
Potenzielle therapeutische SARS-CoV-2-Targets
Pairo-Castineira et al nutzten die Mendelsche Randomisierung, um potenzielle Targets für das Repurposing von zugelassenen Medikamenten zu finden. Eine niedrige Expression von Interferon-alpha/beta-Rezeptor-Beta-Kette (IFNAR2) und eine hohe Expression von TYK2 und C-C-Chemokin-Rezeptor Typ 2 (CCR2) ist mit kritischen Erkrankungen assoziiert. TYK2 ist ein Ziel für JAK-Inhibitoren die bereits in der Medizin eingesetzt werden. Der Rezeptor CCR2 ist in der Lage, das monozytochemotaktische Protein 1 (MCP-1) zu binden. MCP-1-Konzentrationen sind mit einer schwereren Erkrankung assoziiert. Monoklonale Anti-CCR2-Antikörper Therapie in der Behandlung der rheumatoiden Arthritis ist sicher. 6
Dipeptidylpeptidase 9 (DPP9) kodiert eine Serinprotease mit vielfältigen intrazellulären Funktionen, darunter Spaltung des wichtigen antiviralen Signalmediators CXCL10 und Schlüsselrollen Schlüsselrollen bei der Antigenpräsentation und der Aktivierung von Inflammasomen.
Ein weiteres potenzielles therapeutisches Ziel ist PDE-12. Bei Kontakt mit viraler dsDNA produziert OAS1 2'-5'A, das ein Effektorenzym, RNAse L, aktiviert. RNAse L baut doppelsträngige RNA ab, die in der Folge MDA5 aktiviert und zur Interferonproduktion führt. Die Aktivität der endogenen oder exogenen Phosphodiesterase 12 (PDE-12) baut den antiviralen Wirtsmediator 2-5A ab. 6,7
Interferon Antagonism: SARS-CoV-2 Proteine
- (1)
Weitere Ressourcen
Referenzen
- : "Type I and Type III Interferons - Induction, Signaling, Evasion, and Application to Combat COVID-19." in: Cell host & microbe, Vol. 27, Issue 6, pp. 870-878, (2020) (PubMed).
- : "SARS-CoV-2 Disrupts Splicing, Translation, and Protein Trafficking to Suppress Host Defenses." in: Cell, Vol. 183, Issue 5, pp. 1325-1339.e21, (2020) (PubMed).
- : "Imbalanced Host Response to SARS-CoV-2 Drives Development of COVID-19." in: Cell, Vol. 181, Issue 5, pp. 1036-1045.e9, (2020) (PubMed).
- : "Type I Interferon Susceptibility Distinguishes SARS-CoV-2 from SARS-CoV." in: Journal of virology, Vol. 94, Issue 23, (2020) (PubMed).
- : "SARS-CoV-2 ORF3b Is a Potent Interferon Antagonist Whose Activity Is Increased by a Naturally Occurring Elongation Variant." in: Cell reports, Vol. 32, Issue 12, pp. 108185, (2020) (PubMed).
- : "Genetic mechanisms of critical illness in COVID-19." in: Nature, Vol. 591, Issue 7848, pp. 92-98, (2021) (PubMed).
- : "The Role of Phosphodiesterase 12 (PDE12) as a Negative Regulator of the Innate Immune Response and the Discovery of Antiviral Inhibitors." in: The Journal of biological chemistry, Vol. 290, Issue 32, pp. 19681-96, (2015) (PubMed).




