Mutationen des SARS-CoV-2 S Proteins
Genomische Mutationen spielen eine Schlüsselrolle bei der Vermehrung von SARS-COV-2 und von Viren im Allgemeinen. Sie können die Infektion erleichtern und stellen eine zusätzliche Herausforderung für die Erkennung durch die Wirtszelle dar. Dies macht sie zu wichtigen Forschungszielen, insbesondere im Zusammenhang mit dem Design von Impfstoffen und Medikamenten.
- Inkl. N501Y Mutation.
- Quelle: HEK-293 Cells.
- Für ELISA, Ligand Binding Assay.
- Lieferzeit 2-3 Tage.
- Inkl. N501Y, E484K.
- Quelle: HEK-293 Cells
- Für ELISA, Ligand Binding Assay.
- Lieferzeit 2-3 Tage.
- T19R, L452R, T478K, D614G, P681R, D950N
- Quelle: HEK-293 Cells
- Lieferzeit 2-3 Tage.
- Inkl. N501Y, Q498R, H655Y, N679K and P681H.
- Source: HEK-293 Cells
- Für ELISA, Ligand Binding Assay.
- Schnelle Lieferzeit.

- Wildtyp, Full-length Cov-2 Spike Protein.
- Für Assay Development.
- Lieferzeit 2-3 Tage.
Mutationen in B.1.1.7 und in der B.1.351 / 501Y.V2 Linie
Die Mutation N501Y ist eine nichtsynonyme Mutation innerhalb der Rezeptorbindungsdomäne (RBD) des S-Proteins, die von den beiden SARS-CoV-2-Linien B.1.1.7 und 501Y.V2 geteilt wird, die zuerst in Südostengland bzw. Südafrika identifiziert wurden. Es ist einer der Schlüsselkontaktreste innerhalb der RBD und wurde als Erhöhung der Bindungsaffinität zu humanem und murinem ACE2 identifiziert. Derzeit gibt es keine Hinweise darauf, dass die Mutationen in diesen Varianten einen Einfluss auf den Schweregrad der Erkrankung oder die Wirksamkeit des Impfstoffs haben. Es wurde jedoch spekuliert, dass sie zu höheren Konzentrationen in den oberen Atemwegen führen und damit die Übertragungsrate des Virus um etwa 50% erhöhen.
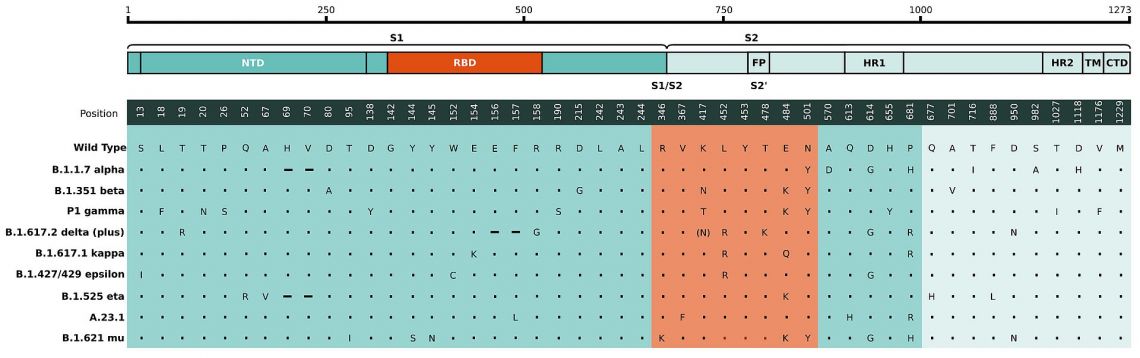
Fig. 1: Aminosäureveränderungen in der Spike-Region der Genome, die auf die Struktur der Spike-Proteinsequenzen abgebildet wurden. Vergleich der in B.1.351 / 501Y.V2 (SA), B.1.1.7 (UK) A.23.1 (UG) und P.1 (BR) identifizierten Mutationen im viralen Spike. RBD-Region ist hervorgehoben (orange).
Die E484K-Mutation, die in den neuen Linien 501Y.S2 und B.1.1.28 aus Südafrika bzw. Brasilien vorhanden ist, betrifft einen Rest innerhalb der RBD, der sich als wichtig für die Bindung vieler neutralisierender Antikörper erwiesen hat. Dementsprechend beeinträchtigt diese Mutation die Antikörpererkennung und ermöglicht die Immunflucht von SARS-CoV-2. Das Virus, das diese Mutation trägt, entgeht nachweislich der Erkennung durch Antikörper in Rekonvaleszentenseren von Menschen und kann somit die Wirksamkeit von Impfstoffen verändern.7,8,9
D614G Mutation
Die Mutation D614G (Asp614Gly) ist in der europäischen Region dominant geworden, was auf einen Fitnessvorteil gegenüber dem ursprünglichen Wuhan-Stamm hinweist, der eine schnellere Verbreitung ermöglicht. Mehrere neuere Studien deuten darauf hin, dass die D614G-Mutation die Interaktion zwischen den S1- und S2-Einheiten verringert, was die Ablösung von S1 von dem an die virale Membran gebundenen S2 erleichtert. Dies führt zu einem Anstieg des gesamten in das Virion eingebauten S-Proteins und somit zu stabileren Viruspartikeln.1,2,3 Wir bieten auch die SARS-CoV-2 S-Protein D614G-Mutante in einer trimeren Form an, was den Forschern detailliertere virologische und immunologische Studien über die biologische Wirkung der Mutation ermöglicht.
- (4)
- (4)
- (4)
Weitere Mutationen des Spike Proteins
Es wurden mehrere weitere Mutationen entdeckt. Die Mutationen V367F (Val367Phe), N354D (Asn354Asp), W436R (Trp436Arg) oder V483A (Val483Ala) des S1-Proteins haben gezeigt, dass sie mit höherer Affinität an ACE2 binden.4,5 Die Mutationen V483A und G476S (Gly476Ser) wurden bereits in der MERS- und SARS-CoV-Forschung im Zusammenhang mit der Rezeptor-Bindungsaffinität beim Menschen beschrieben.10 R408I (Arg408Ile) hingegen reduziert möglicherweise die ACE2-Bindungsaffinität.6
- (1)
- (3)
- (4)
- (1)
- (3)
- (1)
- (3)
Proteinstruktur einiger S-Mutationsproteine
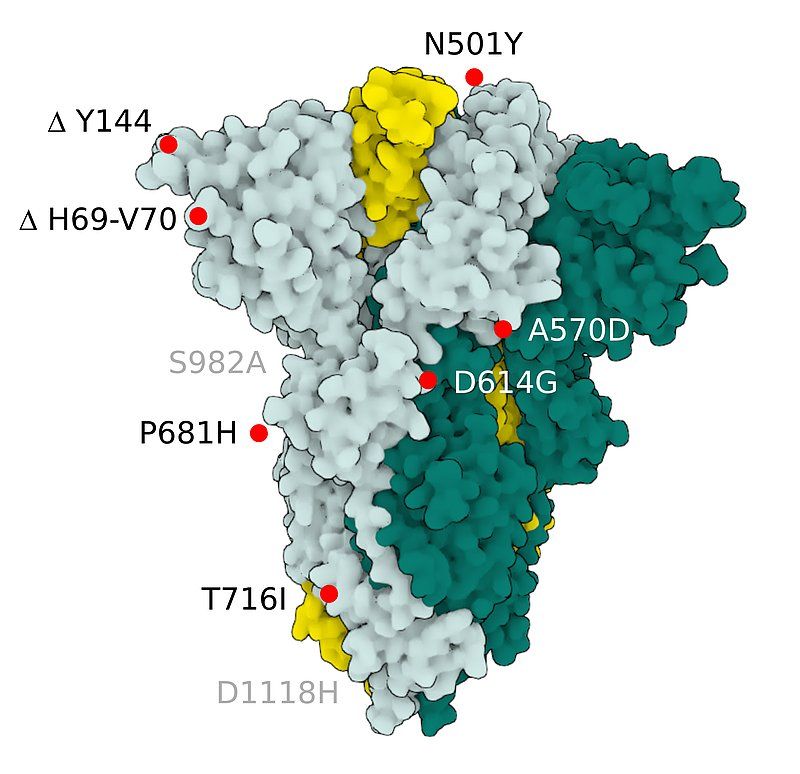
Fig.2 SARS-CoV-2 S Protein B.1.1.7 (alpha)
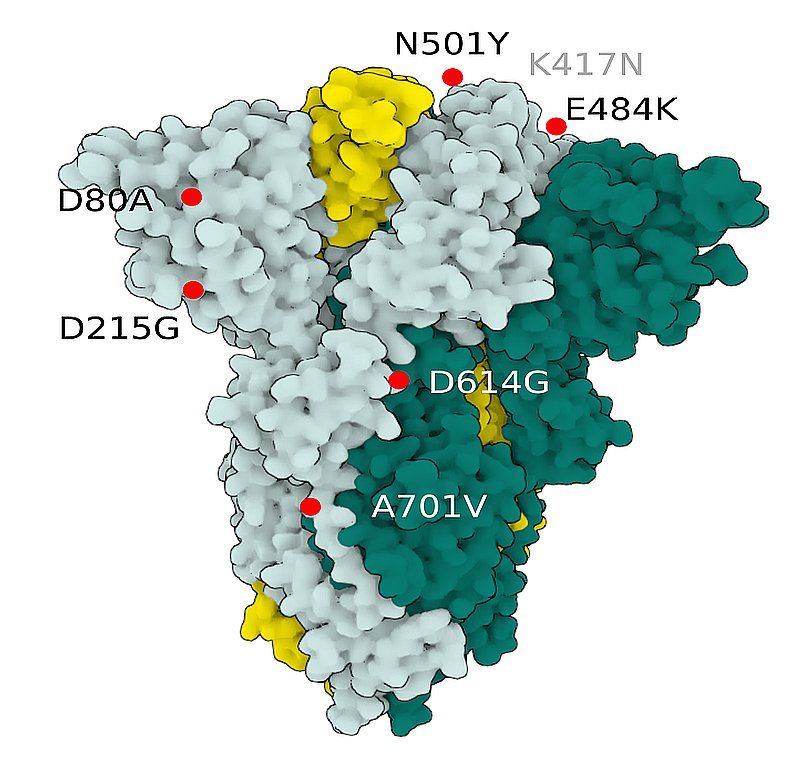
Fig.3 SARS-CoV-2 S Protein B.1.351 (beta)
Fig.4 SARS-CoV-2 S Protein B.1.617.2 (delta)
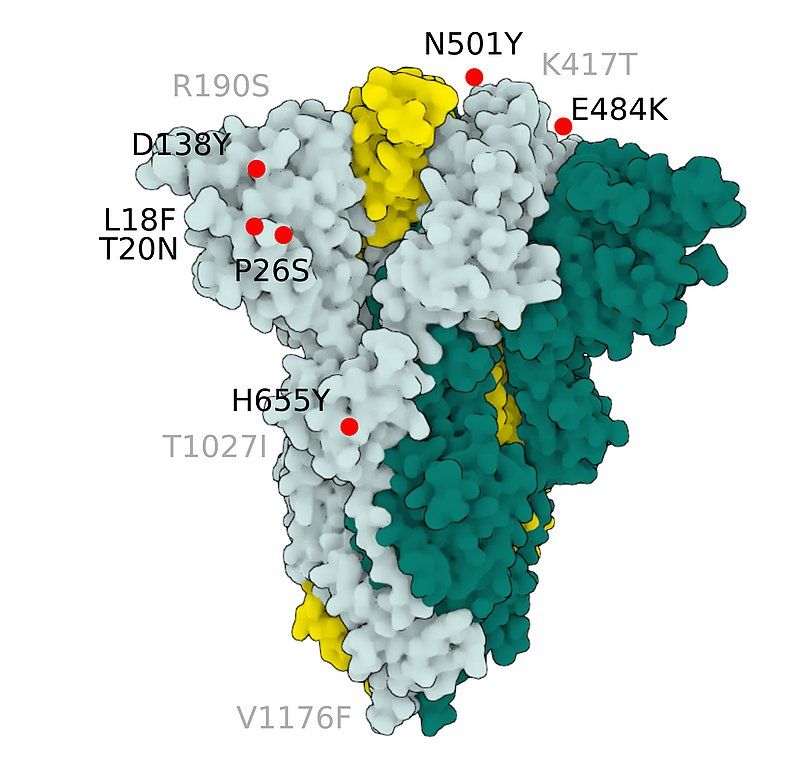
Fig.5 SARS-CoV-2 S Protein P.1 (gamma)
Referenzen
- (1) Jie Hu et al. The D614G mutation of SARS-CoV-2 spike protein enhances viral infectivity and decreases neutralization sensitivity to individual convalescent sera. bioRxviv (2020).
- (2) Korber B. et al.Spike mutation pipeline reveals the emergence of a more transmissible form of SARS-CoV-2. bioRxviv (2020). doi.org/10.1101/2020.04.29.069054.
- (3) Lizhou Zhang et al. The D614G mutation in the SARS-CoV-2 spike protein reduces S1 shedding and increases infectivity. bioRxviv (2020). doi.org/10.1101/2020.06.12.148726.
- (4) Junxian Ou et al. Emergence of RBD mutations in circulating SARS-CoV-2 strains enhancing the structural stability and human ACE2 receptor affinity of the spike protein. bioRxiv (2020). doi:10.1101/2020.03.15.991844v4
- (5) Saha, P. et al.Mutations in Spike Protein of SARS-CoV-2 Modulate Receptor Binding, Membrane Fusion and Immunogenicity: An Insight into Viral Tropism and Pathogenesis of COVID-19. chemRxiv (2020). doi:10.26434/chemrxiv.12320567.v1
- (6) Jian Shang, Yushun Wan, Chuming Luo, Gang Ye, Qibin Geng, Ashley Auerbach, Fang Li. Cell entry mechanisms of SARS-CoV-2. Proceedings of the National Academy of Sciences May 2020, 117 (21) 11727-11734; DOI: 10.1073/pnas.2003138117
- (7) Allison J. Greaney, Andrea N. Loes, Katharine H.D. Crawford, Tyler N. Starr, Keara D. Malone, Helen Y. Chu, Jesse D. Bloom, bioRxiv 2020.12.31.425021; doi: https://doi.org/10.1101/2020.12.31.425021
- (8) Nicholas G. Davies, Rosanna C. Barnard, Christopher I. Jarvis, Adam J. Kucharski, James Munday, Carl A. B. Pearson, Timothy W. Russell, Damien C. Tully, Sam Abbott, Amy Gimma, William Waites, Kerry LM Wong, Kevin van Zandvoort, CMMID COVID-19 Working Group, Rosalind M. Eggo, Sebastian Funk, Mark Jit, Katherine E. Atkins, W. John Edmunds. Estimated transmissibility and severity of novel SARS-CoV-2 Variant of Concern 202012/01 in England. medRxiv 2020.12.24.20248822; doi: https://doi.org/10.1101/2020.12.24.20248822
- (9) Houriiyah Tegally, Eduan Wilkinson, Marta Giovanetti, et al. Emergence and rapid spread of a new severe acute respiratory syndrome-related coronavirus 2 (SARS-CoV-2) lineage with multiple spike mutations in South Africa. medRxiv 2020.12.21.20248640; doi: https://doi.org/10.1101/2020.12.21.20248640
- (10) Kim JS, Jang JH, Kim JM, Chung YS, Yoo CK, Han MG. Genome-Wide Identification and Characterization of Point Mutations in the SARS-CoV-2 Genome. Osong Public Health Res Perspect. 2020;11(3):101-111. doi:10.24171/j.phrp.2020.11.3.05
- (11) Pengfei Wang, Lihong Liu, Sho Iketani, Yang Luo et al. Increased Resistance of SARS-CoV-2 Variants B.1.351 and B.1.1.7 to Antibody Neutralization. bioRxiv preprint posted January 26, 2021. ; https://doi.org/10.1101/2021.01.25.428137doi.




