Exosomen sind extrazelluläre Mikrovesikel (EMV), die von den meisten an der interzellulären Kommunikation beteiligten Zellen ausgeschieden werden. Sie sind in Körperflüssigkeiten vorhanden und können je nach Wirtszelle eine Vielzahl verschiedener Proteine enthalten. Ihr Inhalt wird außerdem durch den Zellzustand wie Stress oder Aktivierung oder Hemmung bestimmter Signalwege moduliert. Dies macht Exosomen zu hervorragenden Biomarkern für Flüssigbiopsien, z. B. bei der Krebsdiagnose.
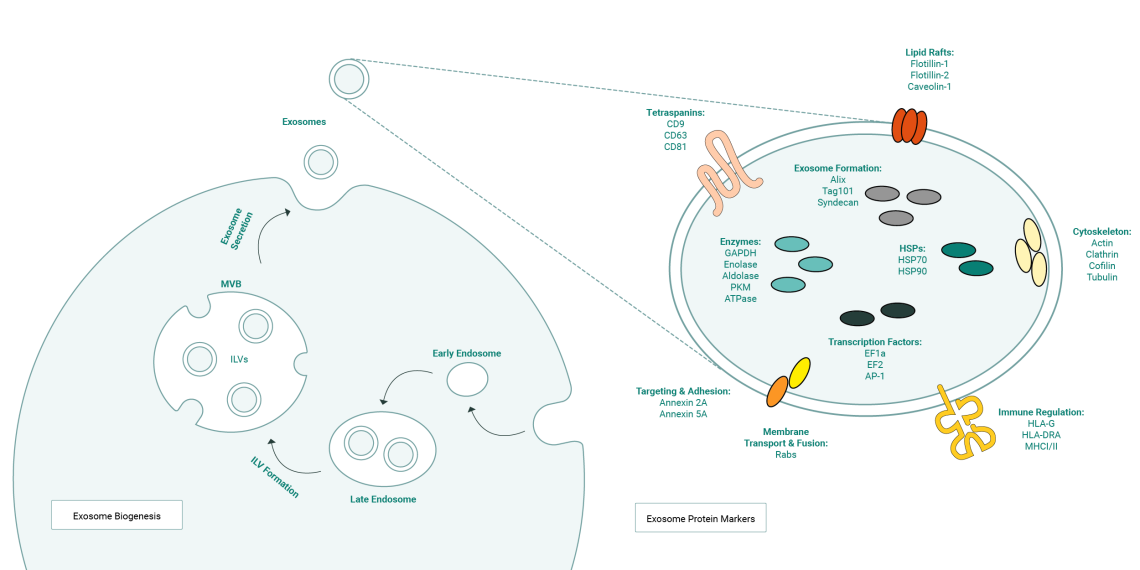
Die Biogenese von Exosomen beginnt mit der Einstülpung von frühen Endosomen. Diese reifen zu späten Endosomen und dann weiter zu multivesikulären Körpern (MVBs). Bei der Bildung von MVBs wird die endosomale Membran nach innen geknotet, was zur Sequestrierung von Zytoplasmakomponenten in intraluminale Vesikel (ILVs) führt. Die ILVs werden durch Exozytose in den extrazellulären Raum freigesetzt, was den Exosomen ihren Namen einbrachte. Exosomen können eine Vielzahl von Exosomen-Proteinmarkern enthalten, je nachdem, welche Wirtszelle sie beherbergen und in welchem Zustand sich die Zelle befindet (z. B. Stress, Aktivierung oder Hemmung bestimmter Signalwege). Tetraspanine wie CD9, CD63 und CD81 sind die häufigsten kanonischen Exosomenmarkerproteine. Durch ihre Oberflächenlokalisierung eignen sich Tetraspanine besonders gut für die Immunmarkierung und Reinigung von Exosomen aus biologischen Proben. Komponenten des für den Transport erforderlichen endosomalen Sortierkomplexes (ESCRT) wie TSG101 und Alix, Zytoskelettproteine, Integrine und Annexine sind ebenfalls auf Exosomen angereichert. Diese Moleküle spielen eine zentrale Rolle bei der Ausrichtung von Exosomen und der Zelladhäsion.
Was sind Exosomen?
Exosomen sind kleine (50-120 nm) aus Endosomen stammende extrazelluläre Mikrovesikel (EMV), die eine entscheidende Rolle bei der interzellulären Kommunikation spielen, indem sie verschiedene Biomoleküle wie Proteine, Lipide und Nukleinsäuren zwischen den Zellen transportieren. Sie wurden erstmals in den frühen 1980er Jahren in den Kulturmedien von Retikulozyten beobachtet. Der Begriff Exosom basiert auf der Beobachtung, dass sie durch Exozytose in das extrazelluläre Medium freigesetzt werden. Exosomen weisen eine ähnliche Topologie wie die Plasmamembran auf und werden von praktisch allen Zelltypen freigesetzt und wurden in allen Körperflüssigkeiten nachgewiesen.
Hol dir unser Exosom Marker Poster!
Wenn Sie auf den unten stehenden Link klicken, können Sie eine Kopie unseres Exosom-Posters im PDF-Format herunterladen.
Download PDFWie entstehen Exosomen?
Die Biogenese von Exosomen beginnt in der Regel mit der Internalisierung von extrazellulärem Material durch Endozytose, was zur Bildung von frühen Endosomen führt, die membrangebundene Organellen sind und die internalisierte Fracht enthalten. Diese reifen dann zu späten Endosomen heran, ein Prozess, der von Veränderungen der Membranzusammensetzung und der Sortierung der Ladung begleitet wird. Späte Endosomen können sich zu MVBs entwickeln, die durch das Vorhandensein von intraluminalen Vesikeln (ILVs) gekennzeichnet sind, die durch Einstülpung der endosomalen Membran gebildet werden. In den MVB werden Proteine, Lipide, Nukleinsäuren und andere Moleküle in ILVs sortiert. Diese Sortierung ist ein entscheidender Schritt in der Exosomenbiogenese und wird durch die für den Transport erforderlichen endosomalen Sortierkomplexe (ESCRT) erreicht. Der ESCRT besteht aus mehreren Proteinkomplexen (ESCRT-0, ESCRT-I, ESCRT-II, ESCRT-III), die zusammenarbeiten, um Ladung zu erkennen und in knospende ILVs einzuschließen. MVBs können dann entweder mit Lysosomen fusionieren, um abgebaut zu werden, oder sie können mit der Plasmamembran fusionieren und die ILVs als Exosomen in den extrazellulären Raum freisetzen.
Wie werden Exosomen Identifiziert?
Exosomen enthalten je nach Wirtszelle eine Vielzahl unterschiedlicher Proteine, und ihre Bestandteile werden durch den Zellzustand (z. B. Stress, Aktivierung oder Hemmung bestimmter Signalwege) weiter moduliert. Tetraspanine wie CD9, CD63 und CD81 sind die häufigsten kanonischen Exosomen-Markerproteine, die auf der Vesikeloberfläche vorhanden sind. Die Oberflächenlokalisierung von Tetraspanin-Antigenen macht sie zu guten Kandidaten für die Immunmarkierung und Reinigung von Exosomen aus biologischen Proben. Komponenten des für den Transport erforderlichen endosomalen Sortierkomplexes (ESCRT) wie TSG101 und Alix, Zytoskelettproteine, Integrine und Annexine sind ebenfalls auf Exosomen angereichert; diese Moleküle spielen eine zentrale Rolle bei der Ausrichtung von Exosomen und der Zelladhäsion.
Warum sind Exosomen wichtig?
Die Sekretion von Exosomen erfolgt konstitutiv, obwohl die Rate der Exosomensekretion und die Zusammensetzung von Exosomen durch eine Vielzahl von intrinsischen oder extrinsischen Faktoren (z. B. Zellstress, Signalkaskaden) erhöht werden kann. Trotz ihrer ubiquitären Natur werden Exosomen als unkonventionelle Bestandteile des sekretorischen Weges angesehen.
Da Exosomen von fast allen Zelltypen sezerniert werden, spiegelt ihre Zusammensetzung die Vielfalt ihrer Wirte wider und hängt stark von der Art der Zelle ab, aus der sie stammen. Ihre molekulare Zusammensetzung spiegelt auch physiologische oder pathophysiologische Veränderungen in ihrer Ursprungszelle oder ihrem Ursprungsgewebe wider. Dies macht Exosomen zu hervorragenden Biomarkern für Flüssigbiopsien zur Diagnose und Verfolgung des Krankheitsverlaufs. Für die Krebsdiagnostik haben Exosomen gegenüber zirkulierenden Tumorzellen (CTCs) oder zirkulierender Tumor-DNA (ctDNA) aufgrund ihrer Häufigkeit, Stabilität und der großen Vielfalt der enthaltenen Markermoleküle erhebliche Vorteile. Exosomen sind auch an der Zell-Zell-Kommunikation beteiligt. Exosomenkomponenten können direkt auf benachbarte Zellen übertragen werden oder durch verschiedene Zellen wandern, bevor sie über eine als Transzytose bekannte Methode ihr Ziel erreichen. Auf diese Weise können Exosomen Signale über große Entfernungen übertragen, bei denen eine einfache Diffusion unzureichend sein kann. Ihre Rolle bei der Zell-Zell-Kommunikation lässt vermuten, dass Exosomen bei vielen physiologischen Prozessen eine tiefere Rolle spielen. Diese Hypothese wird durch die Beobachtung gestützt, dass die Exosomen-Signalübertragung eine direkte Rolle bei der Entwicklung und Musterbildung, der Immunantwort, der neuronalen Kommunikation und der Gewebereparatur spielt. Bei einigen Pathologien fungieren Exosomen auch als Vektoren; von Tumorzellen stammende Exosomen spielen eine aktive Rolle bei der Angiogenese und Metastasierung von Tumoren. Exosomen, die von stimulierten Blutzellen und dem Gefäßendothel abgegeben werden, sind an neurologischen Erkrankungen wie Multipler Sklerose, transitorischen ischämischen Attacken und dem Antiphospholipid-Syndrom beteiligt. Exosomen können auch beschädigtes Zellmaterial transportieren. Sie erleichtern die Verbreitung toxischer Formen aggregierter Proteine wie α-Synuclein und β-Amyloid und tragen zum Fortschreiten neurodegenerativer Erkrankungen bei. Einige Forschungsarbeiten deuten auch darauf hin, dass der Exosomen-Transport von viralen Krankheitserregern wie SARS-CoV-2 ausgenutzt wird, um zwischen den Wirtszellen zu wandern und der Immunerkennung zu entgehen. Aufgrund ihrer geringen Größe und einfachen Struktur können Exosomen manchmal die Blut-Hirn-Schranke überwinden. Es wurde vorgeschlagen, dass Exosomen als Verabreichungssystem für das zentrale Nervensystem dienen könnten, um neuropathische Krankheiten ohne invasive Eingriffe zu behandeln. Die Verwendung von Exosomen zur Übertragung genetischer Informationen oder zur Verabreichung von therapeutischen Wirkstoffen ist ein derzeit noch wenig erforschtes Gebiet, das ein großes medizinisches Potenzial birgt.Wie untersucht man Exosomen?
Das Exosom-Sekretom ist groß und vielfältig und enthält viele verschiedene Marker (siehe http://www.exocarta.org/). Das Vorhandensein von kanonischen Oberflächenmarkern wie den oben genannten ermöglicht die Reinigung und eingehende Untersuchung der Exosomensekretion und des Exosomeninhalts aus verschiedenen Probentypen.
antibodies-online bietet eine Reihe von Antikörpern und ELISA-Kits für den Nachweis von bekannten Exosomen-Proteinen an.
Exosome Marker Antikörper
Aus der jüngsten Literatur geht hervor, dass zu den wichtigsten Protein-Exosom-Markern folgende gehören:
| Protein | Gen | GeneID | Uniprot | Ref | exocarta Top 100 Proteine | TS | EF | LP | TA | CS | AG | MT | AP | HS | EN | RG | CA | II | VI | ND |
| 14-3-3 protein epsilon | YWHAE | 7531 | P62258 | - | 22 | X | ||||||||||||||
| 14-3-3 protein beta/alpha | YWHAB | 7529 | P31946 | 50 | x | |||||||||||||||
| 14-3-3 protein eta | YWHAH | 7533 | Q04917 | 94 | x | |||||||||||||||
| 14-3-3 protein gamma | YWHAG | 7532 | P61981 | 54 | x | |||||||||||||||
| 14-3-3 protein theta | YWHAQ | 10971 | P27348 | 56 | x | |||||||||||||||
| 14-3-3 protein zeta/delta | YWHAZ | 7534 | P63104 | - | 15 | X | ||||||||||||||
| 78 kDa glucose-regulated protein | HSPA5 | 3309 | P11021 | (1) | 35 | X | ||||||||||||||
| Actin, cytoplasmic 1 | ACTB | 60 | P60709 | - | 5 | X | ||||||||||||||
| ADAM10 | ADAM10 | 102 | O14672 | (2) | - | X | X | X | X | |||||||||||
| Alix | PDCD6IP | 10015 | Q8WUM4 | (3) | 2 | X | ||||||||||||||
| Alpha-Enolase | ENO1 | 2023 | P06733 | (4) | 9 | X | ||||||||||||||
| Alpha-Synclein | SNCA | 6622 | P37840 | (32) | - | X | X | |||||||||||||
| Aminopeptidase N | ANPEP | 290 | P15144 | - | - | X | X | |||||||||||||
| Beta-amyloid | APP | 351 | P05067 | (5) | - | X | ||||||||||||||
| Annexin A1 | ANXA1 | 301 | P04083 | 53 | x | |||||||||||||||
| Annexin A11 | ANXA11 | 311 | P50995 | 68 | x | |||||||||||||||
| Annexin A4 | ANXA4 | 307 | P09525 | 72 | x | |||||||||||||||
| Annexin A6 | ANXA6 | 309 | P08133 | 67 | x | |||||||||||||||
| Annexin A5 | ANXA5 | 308 | P08758 | (6) | 20 | X | X | |||||||||||||
| Annexin A2 | ANXA2 | 302 | P07355 | (7) | 6 | X | ||||||||||||||
| AP-1 | JUN | 3725 | P05412 | - | - | X | X | |||||||||||||
| ATP citrate lyase | ACLY | 47 | P53396 | - | 72 | X | ||||||||||||||
| ATPase | ATP1A1 | 476 | P05023 | - | 39 | X | ||||||||||||||
| Basigin | BSG | 682 | P35613 | (8) | - | X | X | |||||||||||||
| Caveolin-1 | CAV1 | 857 | Q03135 | (9), (10) | - | X | X | |||||||||||||
| CD9 | CD9 | 928 | P21926 | (11) | 1 | X | ||||||||||||||
| CD11a | ITGAL | 3683 | P20701 | (12) | - | X | X | |||||||||||||
| CD11b | ITGAX | 3687 | P11215 | (12) | - | X | X | |||||||||||||
| CD11c | ITGAM | 3684 | P20702 | (12) | - | X | X | |||||||||||||
| CD29 | ITGB1 | 3688 | P05556 | (12) | 34 | X | X | |||||||||||||
| CD37 | CD37 | 951 | P11049 | (11) | - | X | ||||||||||||||
| CD44 | CD44 | 960 | P16070 | (13) | - | X | X | |||||||||||||
| CD49f | ITGA6 | 3655 | P23229 | (12) | 89 | X | X | |||||||||||||
| CD53 | CD53 | 963 | P19397 | - | x | |||||||||||||||
| CD63 | CD63 | 967 | P08962 | (10), (11) | 7 | X | X | |||||||||||||
| CD81 | CD81 | 975 | P60033 | (14) | 24 | X | X | |||||||||||||
| CD82 | CD82 | 3732 | P27701 | (11) | - | X | ||||||||||||||
| CD142 | TF | 2152 | P13726 | (15) | - | X | X | |||||||||||||
| CD146 | MCAM | 4162 | P43121 | (15) | - | X | X | |||||||||||||
| CD163 | CD163 | 9332 | Q86VB7 | (15) | - | X | X | X | ||||||||||||
| Clathrin heavy chain 1 | CLTC | 1213 | Q00610 | - | 23 | X | ||||||||||||||
| Claudin-1 | CLDN1 | 9076 | O95832 | (8) | - | X | ||||||||||||||
| Cofilin-1 | CFL1 | 1072 | P23528 | - | 25 | X | ||||||||||||||
| - | - | (16) | - | X | ||||||||||||||||
| - | - | (16) | - | X | ||||||||||||||||
| EF-1-alpha-1 | EEF1A1 | 1915 | P68104 | (4) | 14 | X | ||||||||||||||
| EF2 | EEF2 | 1938 | P13639 | - | 17 | X | ||||||||||||||
| EGFR | EGFR | 1956 | P00533 | (15) | - | X | ||||||||||||||
| Ep-CAM | EPCAM | 4072 | P16422 | (17), (18) | - | X | X | |||||||||||||
| Fatty acid synthase | FASN | 2194 | P49327 | (3) | 21 | X | X | |||||||||||||
| Fibronectin | FN1 | 2335 | P02751 | 93 | ||||||||||||||||
| Flotillin-1 | FLOT1 | 10211 | O75955 | (18), (19) | 41 | X | X | |||||||||||||
| Flotillin-2 | FLOT2 | 2319 | Q14254 | (19) | - | X | ||||||||||||||
| Fructose-bisphosphate aldolase A | ALDOA | 226 | P04075 | - | 18 | X | ||||||||||||||
| Gelsolin | GSN | 2934 | P06396 | 92 | x | x | ||||||||||||||
| Glyceraldehyde-3-phosphate dehydrogenase | GAPDH | 2597 | P04406 | - | 4 | X | ||||||||||||||
| HCV core protein | - | - | (20) | - | X | |||||||||||||||
| Heat shock 70 kDa protein 1A | HSPA1A | 3303 | P0DMV8 | 51 | x | |||||||||||||||
| Heat shock protein HSP 90-alpha | HSP90AA1 | 3320 | P07900 | (1) | 10 | X | X | |||||||||||||
| Heat shock protein HSP 90-beta | HSP90AB1 | 3326 | P08238 | (1) | 19 | X | ||||||||||||||
| Heparanase | HPSE | 10855 | Q9Y251 | (21) | - | X | X | |||||||||||||
| - | - | (20) | - | X | ||||||||||||||||
| - | - | (20) | - | X | ||||||||||||||||
| HLA-DRA | HLA-DRA | 3122 | P01903 | (22) | - | X | X | X | ||||||||||||
| HLA-G | 3135 | P17693 | (23) | - | X | X | X | |||||||||||||
| Hsc70 | HSPA8 | 3312 | P11142 | (1) | 3 | X | ||||||||||||||
| - | - | (16) | - | X | ||||||||||||||||
| Tax | - | - | (20) | - | X | |||||||||||||||
| Huntingtin | HTT | 3064 | P42858 | (5) | - | X | X | |||||||||||||
| ICAM-1 | ICAM1 | 3383 | P05362 | (24) | - | X | ||||||||||||||
| Leucine-rich receptor kinase 2 | LRRK2 | 120892 | Q5S007 | (5) | - | X | ||||||||||||||
| L-lactate dehydrogenase A chain | LDHA | 3939 | P00338 | - | 13 | X | ||||||||||||||
| Lysosome-associated membrane glycoprotein 1 | LAMP1 | 3916 | P11279 | (25) | - | X | X | |||||||||||||
| Lysosome-associated membrane glycoprotein 2 | LAMP2 | 3920 | P13473 | (23) | 88 | X | X | |||||||||||||
| MHCI | - | - | (26) | - | X | X | ||||||||||||||
| MHCII | - | - | (26) | - | X | X | ||||||||||||||
| MUC1 | MUC1 | 4582 | P15941 | (15) | - | X | X | |||||||||||||
| N-cadherin | CDH2 | 1000 | P19022 | (15) | - | X | X | |||||||||||||
| Phosphoglycerate kinase 1 | PGK1 | 5230 | P00558 | (4) | 16 | X | ||||||||||||||
| Placental Alkaline Phosphatase | ALPP | 250 | P05187 | (15) | - | X | X | |||||||||||||
| Prion proteins | - | - | (5) | - | X | |||||||||||||||
| Prostate-specific antigen | KLK3 | 354 | P07288 | (27) | - | X | X | |||||||||||||
| Pyruvate kinase PKM | PKM | 5315 | P14618 | (28) | 12 | X | X | |||||||||||||
| Rab-14 | RAB14 | 51552 | P61106 | - | 75 | X | ||||||||||||||
| Rab-5a | RAB5A | 5868 | P20339 | - | 80 | X | ||||||||||||||
| Rab-5b | RAB5B | 5869 | P61020 | - | 86 | X | ||||||||||||||
| Rab-5c | RAB5C | 5878 | P51148 | - | 64 | X | ||||||||||||||
| Rab-7 | RAB7A | 7879 | P51149 | - | 61 | X | ||||||||||||||
| Rap 1B | RAP1B | 5908 | P61224 | - | 33 | X | ||||||||||||||
| Superoxide dismutase | SOD1 | 6647 | P00441 | - | x | x | ||||||||||||||
| Syndecan-1 | SDC1 | 6382 | P18827 | (29) | - | X | ||||||||||||||
| Syndecan-4 | SDC4 | 6385 | P31431 | (29) | - | X | ||||||||||||||
| Syntenin-1 | SDCBP | 6386 | O00560 | (30) | 8 | X | ||||||||||||||
| TARDBP | TDP-43 | 23435 | Q13148 | - | x | |||||||||||||||
| Transitional endoplasmic reticulum ATPase | VCP | 7415 | P55072 | 26 | ||||||||||||||||
| Triosephosphate isomerase | TPI1 | 7167 | P60174 | 27 | x | |||||||||||||||
| Tumor-Associated Glycoprotein | TAG-72 | - | - | (15) | - | X | ||||||||||||||
| Tetraspanin-8 | Tspan8 | 7103 | P19075 | (15) | - | X | X | |||||||||||||
| Tsg101 | TSG101 | 7251 | Q99816 | (31) | 11 | X | ||||||||||||||
| Tubulin alpha-1C chain | TUBA1C | 84790 | Q9BQE3 | - | x | |||||||||||||||
| Tubulin alpha-4A chain | TUBA4A | 7277 | P68366 | - | x | |||||||||||||||
| Tubulin beta-2B chain | TUBB2B | 347733 | Q9BVA1 | - | x | |||||||||||||||
| Tubulin beta-4B chain | TUBB4B | 10383 | P68371 | - | x | |||||||||||||||
| Vacuolar-sorting protein 35 | VPS35 | 55737 | Q96QK1 | (5) | - | X | X |

Hol dir unser Exosom Marker Poster!
Wenn Sie auf den unten stehenden Link klicken, können Sie eine Kopie unseres Exosom-Posters im PDF-Format herunterladen.
Download PDFReferenzen
- : "ICAM-1 on exosomes from mature dendritic cells is critical for efficient naive T-cell priming." in: Blood, Vol. 106, Issue 1, pp. 216-23, (2005) (PubMed).
- : "Exosome-dependent trafficking of HSP70: a novel secretory pathway for cellular stress proteins." in: The Journal of biological chemistry, Vol. 280, Issue 24, pp. 23349-55, (2005) (PubMed).
- : "The regulation of exosome secretion: a novel function of the p53 protein." in: Cancer research, Vol. 66, Issue 9, pp. 4795-801, (2006) (PubMed).
- : "B cell activation regulates exosomal HLA production." in: European journal of immunology, Vol. 38, Issue 5, pp. 1423-34, (2008) (PubMed).
- : "CD44 and EpCAM: cancer-initiating cell markers." in: Current molecular medicine, Vol. 8, Issue 8, pp. 784-804, (2009) (PubMed).
- : "High levels of exosomes expressing CD63 and caveolin-1 in plasma of melanoma patients." in: PloS one, Vol. 4, Issue 4, pp. e5219, (2009) (PubMed).
- : "Claudin-containing exosomes in the peripheral circulation of women with ovarian cancer." in: BMC cancer, Vol. 9, pp. 244, (2009) (PubMed).
- : "Lipid raft endocytosis and exosomal transport facilitate extracellular trafficking of annexin A2." in: The Journal of biological chemistry, Vol. 286, Issue 35, pp. 30911-30925, (2011) (PubMed).
- : "Microvesicles and viral infection." in: Journal of virology, Vol. 85, Issue 24, pp. 12844-54, (2012) (PubMed).
- : "The roles of flotillin microdomains--endocytosis and beyond." in: Journal of cell science, Vol. 124, Issue Pt 23, pp. 3933-40, (2012) (PubMed).
- : "Soluble serum CD81 is elevated in patients with chronic hepatitis C and correlates with alanine aminotransferase serum activity." in: PloS one, Vol. 7, Issue 2, pp. e30796, (2012) (PubMed).
- : "Syndecan-syntenin-ALIX regulates the biogenesis of exosomes." in: Nature cell biology, Vol. 14, Issue 7, pp. 677-85, (2012) (PubMed).
- : "Quantitative proteomic analysis of exosomes from HIV-1-infected lymphocytic cells." in: Proteomics, Vol. 12, Issue 13, pp. 2203-11, (2012) (PubMed).
- : "Exosomes and microvesicles: extracellular vesicles for genetic information transfer and gene therapy." in: Human molecular genetics, Vol. 21, Issue R1, pp. R125-34, (2013) (PubMed).
- : "Innate immune response of human alveolar type II cells infected with severe acute respiratory syndrome-coronavirus." in: American journal of respiratory cell and molecular biology, Vol. 48, Issue 6, pp. 742-8, (2013) (PubMed).
- : "Heparanase regulates secretion, composition, and function of tumor cell-derived exosomes." in: The Journal of biological chemistry, Vol. 288, Issue 14, pp. 10093-10099, (2013) (PubMed).
- : "Exosome uptake depends on ERK1/2-heat shock protein 27 signaling and lipid Raft-mediated endocytosis negatively regulated by caveolin-1." in: The Journal of biological chemistry, Vol. 288, Issue 24, pp. 17713-24, (2013) (PubMed).
- : "Proteomic profiling of exosomes leads to the identification of novel biomarkers for prostate cancer." in: PloS one, Vol. 8, Issue 12, pp. e82589, (2014) (PubMed).
- : "Syntenin-ALIX exosome biogenesis and budding into multivesicular bodies are controlled by ARF6 and PLD2." in: Nature communications, Vol. 5, pp. 3477, (2014) (PubMed).
- : "Plasma exosomal α-synuclein is likely CNS-derived and increased in Parkinson's disease." in: Acta neuropathologica, Vol. 128, Issue 5, pp. 639-650, (2015) (PubMed).
- : "Tetraspanins in extracellular vesicle formation and function." in: Frontiers in immunology, Vol. 5, pp. 442, (2014) (PubMed).
- : "Exosomes secreted by cortical neurons upon glutamatergic synapse activation specifically interact with neurons." in: Journal of extracellular vesicles, Vol. 3, pp. 24722, (2014) (PubMed).
- : "Antigen Presentation by MHC-Dressed Cells." in: Frontiers in immunology, Vol. 5, pp. 672, (2015) (PubMed).
- : "Anti-cancer fatty-acid derivative induces autophagic cell death through modulation of PKM isoform expression profile mediated by bcr-abl in chronic myeloid leukemia." in: Cancer letters, Vol. 360, Issue 1, pp. 28-38, (2015) (PubMed).
- : "Exosomes: novel biomarkers for clinical diagnosis." in: TheScientificWorldJournal, Vol. 2015, pp. 657086, (2016) (PubMed).
- : "Exosomal proteins as potential diagnostic markers in advanced non-small cell lung carcinoma." in: Journal of extracellular vesicles, Vol. 4, pp. 26659, (2015) (PubMed).
- : "HLA-G: An Immune Checkpoint Molecule." in: Advances in immunology, Vol. 127, pp. 33-144, (2015) (PubMed).
- : "Exosomes and Their Role in the Life Cycle and Pathogenesis of RNA Viruses." in: Viruses, Vol. 7, Issue 6, pp. 3204-25, (2016) (PubMed).
- : "ExoCarta: A Web-Based Compendium of Exosomal Cargo." in: Journal of molecular biology, Vol. 428, Issue 4, pp. 688-692, (2016) (PubMed).
- : "Tumour exosome integrins determine organotropic metastasis. ..." in: Nature, Vol. 527, Issue 7578, pp. 329-35, (2015) (PubMed).
- : "Exosomes in neurological disease, neuroprotection, repair and therapeutics: problems and perspectives." in: Neural regeneration research, Vol. 10, Issue 10, pp. 1565-7, (2015) (PubMed).
- : "Interrogating Circulating Microsomes and Exosomes Using Metal Nanoparticles." in: Small (Weinheim an der Bergstrasse, Germany), Vol. 12, Issue 6, pp. 727-32, (2016) (PubMed).
- : "PSA and beyond: alternative prostate cancer biomarkers." in: Cellular oncology (Dordrecht), Vol. 39, Issue 2, pp. 97-106, (2016) (PubMed).
- : "A Perspective on Extracellular Vesicles Proteomics." in: Frontiers in chemistry, Vol. 5, pp. 102, (2017) (PubMed).
- : "Reassessment of Exosome Composition." in: Cell, Vol. 177, Issue 2, pp. 428-445.e18, (2019) (PubMed).
- : "On the potential role of exosomes in the COVID-19 reinfection/reactivation opportunity." in: Journal of biomolecular structure & dynamics, Vol. 39, Issue 15, pp. 5831-5842, (2021) (PubMed).
- : "Ultrastructural Evidence for Direct Renal Infection with SARS-CoV-2." in: Journal of the American Society of Nephrology : JASN, Vol. 31, Issue 8, pp. 1683-1687, (2020) (PubMed).
- : "Extracellular vesicles and amyotrophic lateral sclerosis: from misfolded protein vehicles to promising clinical biomarkers." in: Cellular and molecular life sciences : CMLS, Vol. 78, Issue 2, pp. 561-572, (2021) (PubMed).
- : "The exosome journey: from biogenesis to uptake and intracellular signalling." in: Cell communication and signaling : CCS, Vol. 19, Issue 1, pp. 47, (2022) (PubMed).
- : "Emerging Role of Exosomes in Liquid Biopsy for Monitoring Prostate Cancer Invasion and Metastasis." in: Frontiers in cell and developmental biology, Vol. 9, pp. 679527, (2021) (PubMed).
- : "Valosin-Containing Protein (VCP)/p97: A Prognostic Biomarker and Therapeutic Target in Cancer." in: International journal of molecular sciences, Vol. 22, Issue 18, (2021) (PubMed).
- : "Exosomes as a new frontier of cancer liquid biopsy." in: Molecular cancer, Vol. 21, Issue 1, pp. 56, (2022) (PubMed).
- : "Gelsolin: A comprehensive pan-cancer analysis of potential prognosis, diagnostic, and immune biomarkers." in: Frontiers in genetics, Vol. 14, pp. 1093163, (2023) (PubMed).
- : "Vesiclepedia 2024: an extracellular vesicles and extracellular particles repository." in: Nucleic acids research, Vol. 52, Issue D1, pp. D1694-D1698, (2024) (PubMed).
- : "Roles of exosomes in immunotherapy for solid cancers." in: Cell death & disease, Vol. 15, Issue 2, pp. 106, (2024) (PubMed).





